[研究成果] 有田班と中西班、古関班の共同研究がNat Commun誌に掲載されました!
2022.11.24
「クライオ電顕によるDNMT1の新規の酵素活性化機構の解明」
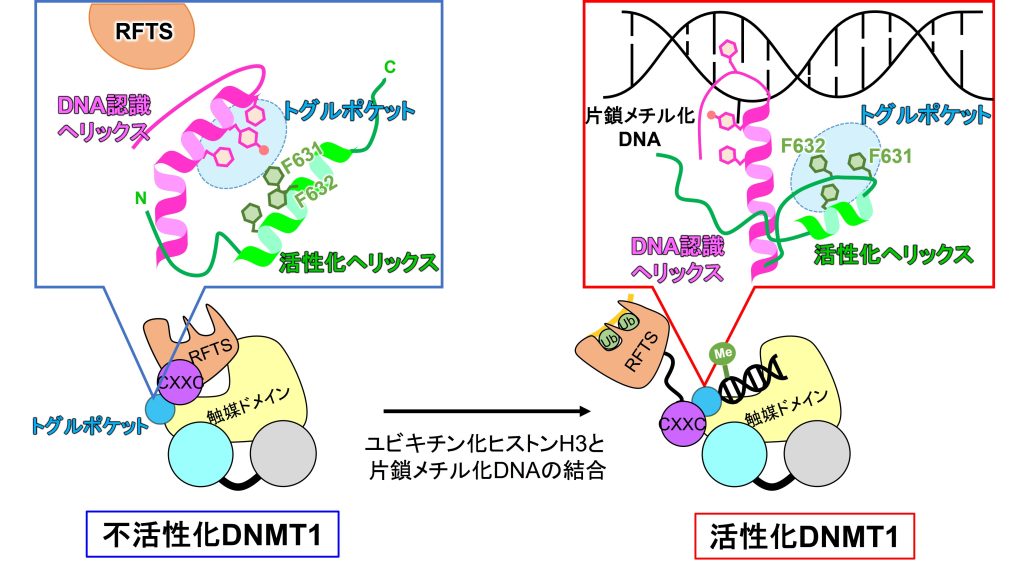
哺乳類のCpG配列中のシトシンのメチル化は、レトロトランスポゾンのサイレンシング、X染色体の不活性化、ゲノムインプリンティングなどの生命現象に関与します。一旦細胞が獲得したDNAメチル化は、細胞形質の維持のために生涯を通して正確に受け継がれます。このDNA維持メチル化には、DNMT1とその呼び込み因子UHRF1が必須です。DNA複製後に一過的に生じたヘミメチル化DNAをUHRF1が認識し、近傍のヌクレオソーム中のヒストンH3をユビキチン化します。DNMT1のRFTSがユビキチン化H3を認識し、DNMT1はヘミメチル化DNAに呼び込まれると同時に、活性化されます。DNMT1は1616アミノ酸残基から成り、N末端からRFTS, CXXC, BAH1, BAH2, 触媒ドメインで構成されます。これまでの構造生物学的な研究から、単体のDNMT1はRFTSドメインが触媒ドメインのDNA結合部位に入り込み自己阻害構造をとることが知られています (図左)。我々はユビキチン化H3の結合によるDNMT1の活性化機構の解明のために、DNMT1とユビキチン化H3とヘミメチル化DNAの3者複合体構造を2.5 Å分解能で決定しました。
得られた構造では、RFTSドメインは完全に触媒ドメインから解離し(構造中ではdisorder)、自己阻害構造が解除されていました。さらに、触媒ドメインの中に活性のオン・オフをスイッチするトグルポケットを発見しました。DNMT1の触媒ドメインにはDNA認識ヘリックスがあります。不活性化型DNMT1では、トグルポケットにDNA認識ヘリックス中のフェニルアラニン残基が入り込んでいることがわかっています。これによりDNA認識ヘリックスは折れ曲がったコンフォメーションをとり、DNA結合が阻害されています (図左)。一方で活性化型DNMT1では、N末端にある活性化へリックス中のF631/F632がトグルポケットに入り込んでいました。これにより、DNA認識ヘリックスは伸びたコンフォメーションを取り、DNAに結合できることがわかりました (図右)。F631/F632のアラニン変異体を用いたin vitroメチル化実験で、変異体DNMT1の活性が消失することがわかりました。さらに、AIDシステムで内在性DNMT1の除去した大腸がん細胞株HCT116で変異体DNMT1を発現させると、ゲノム全体のDNAメチル化レベルを維持できないことがわかりました。これらの実験から今回発見したDNMT1の活性化へリックスは、DNMT1の新規の活性化モチーフであることがわかりました。
活性化へリックス中のF631/F632は脊椎動物で高度に保存されており、今回発見したDNMT1の活性化機構は種を超えた分子メカニズムであることが考えられます。本研究によりDNMT1が多彩で複雑な制御を受けて活性化に至ることがわかり、非ゲノム情報であるDNAメチル化維持の堅牢性の分子機構が明らかになりました。