研究課題名
クロマチン構造と遺伝子発現を接続する一細胞時系列モデリング
研究内容
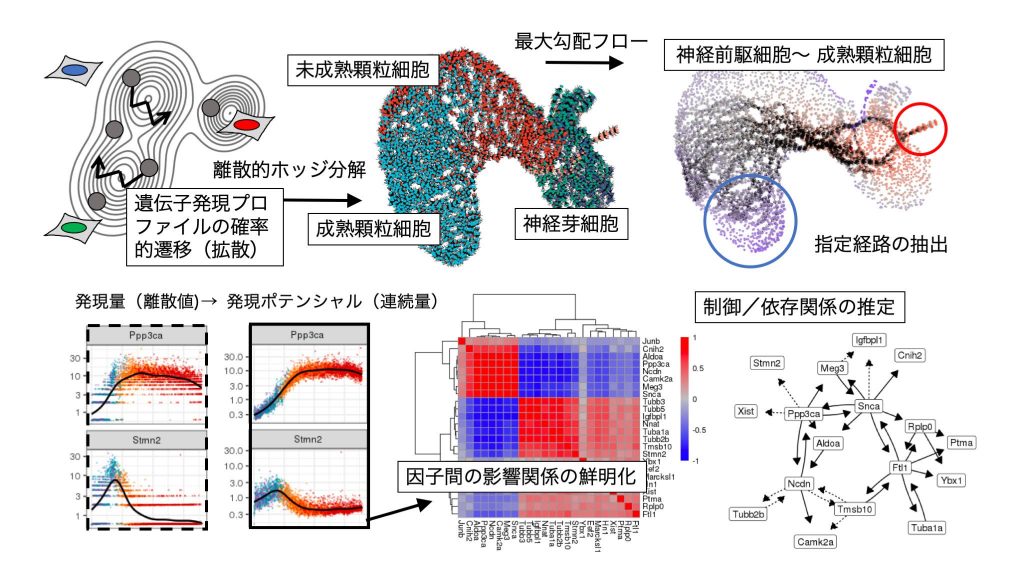
細胞分化は遺伝情報の選択的な獲得により制御されます。その制御は、転写制御因子がゲノム上の制御配列に結合することで行われ、これまで数多くの組み合わせが同定されてきました。ゲノム機能は、分化刺激への応答により惹起される多数分子の組み合わせによってクロマチン構造を変化させ、転写を活性化あるいは抑制します。私はこれまで、多数分子の織りなす転写制御の複雑さを紐解く鍵が、クロマチン構造変化にあると考え、計算機を用い、クロマチン構造を構成するヒストンバリアントの網羅的同定と機能解析を行ってきました(論文4,5)。これまでに、マウス骨格筋幹細胞に発現するヒストンバリアントH3mm7を同定し、H3mm7のクロマチンへの取り込みが、分化に伴う遺伝子発現量の「レート(倍率)変化」を引き起こし、骨格筋再生を制御することを報告しました(論文3)。こうした空間・時間的に限られた局面ではたらく組織特異的な幹細胞の機能を理解するには、組織や単一細胞レベルでヒストンの局在と、その変化がもたらす遺伝子発現のダイナミクスを同時に知る必要があると考えました。そこで本計画では、独自に開発した少数細胞エピゲノム解析技術ChIL法(論文2)によるマルチオミクスデータの取得、およびその解析を行います。さらに、クロマチン構成因子と遺伝子発現制御の制御/依存関係の理解を目指し、現在開発中の単一細胞データの時間構造推定法(論文1)を応用した、新たな遺伝子発現の時系列解析法の確立を目指します。具体的には、単一細胞遺伝子発現データの時系列モデリングを行い、遺伝子発現のみによって説明できない制御構造の変化や、計測対象外の因子による外乱の有無を評価します。ダイナミクスの適切な記述に必須のモデル外部の要素(非ゲノム因子としてのクロマチン構造)の機能を解明することで、クロマチン構造変化による遺伝子発現の新たな理解を目指します。
主な論文
- Maehara K and *Ohkawa Y(2人中1番目)Modeling latent flows on single-cell data using the Hodge decomposition. BioRxiv (2019; preprint)
- †Harada A, †Maehara K, †Handa T, et al.(10人中1番目; co-first)A chromatin integration labelling method enables epigenomic profiling with lower input. Nature Cell Biology 21(2):287-296 (2019)
- †Harada A, †Maehara K et al. (17人中1番目; co-first)Histone H3.3 sub-variant H3mm7 is required for normal skeletal muscle regeneration. Nature Communications 9(1):1400 (2018)
- Maehara K and *Ohkawa Y(2人中1番目)Exploration of nucleosome positioning patterns in transcription factor function. Scientific Reports 6:19620 (2016)
- †Maehara K, †Harada A, et al. (7人中1番目)Tissue-specific expression of histone H3 variants diversified after species separation. Epigenetics & Chromatin, 8:35, (2015)
