研究課題名
非ゲノム複製の破綻がもたらすDNA損傷メカニズムの解明
研究内容
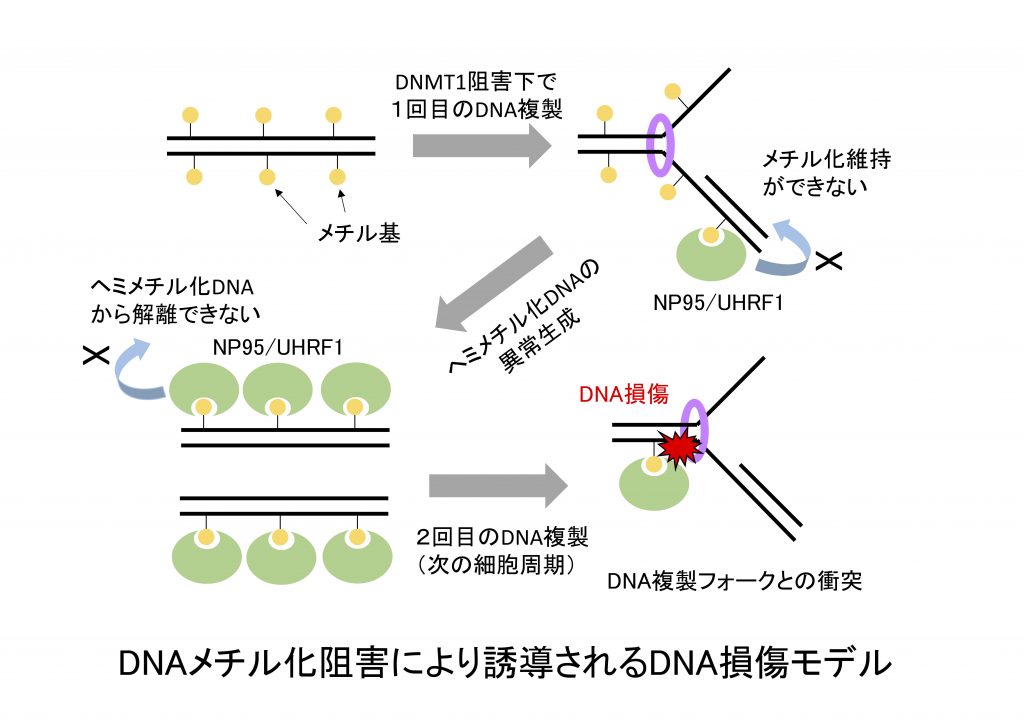
多くの非ゲノム情報の中でもDNAメチル化に関しては継承メカニズムの解明が進んでおり,その過程に関わる多くの因子がこれまで同定されています。具体的には,ゲノム複製の際に生じたヘミメチル化DNA(親鎖にメチル基が付加した状態であるが,新生鎖は修飾されていない状態のDNA)が,DNMT1維持メチル化酵素の働きによって両鎖がメチル化された状態に変換され,親鎖のメチル化パターンが次世代に正確にコピーされることがすでに明らかになっています。私たちは, NP95/UHRF1タンパク質がゲノム複製によって一時的に生じたヘミメチル化DNAに結合し,そこにDNMT1酵素を呼び込むことでメチル化パターンの維持を行なっていることを報告するなど,そのメカニズム解明に貢献してきました。興味深いことに,DNMT1酵素を欠損させた細胞では急激な増殖停止や細胞死が誘導されるという現象が報告されています。遺伝子発現の制御不全だけでは説明できない現象が起こっており,そのメカニズム解明は非ゲノム複製の生物学的意義の再評価に繋がると期待されます。最近,私たちは,DNMT1阻害によって異常に生じたヘミメチル化DNAがDNA損傷の原因になっていること示唆するデータを得ています。また,この損傷はDNMT1阻害を行った最初のS期では誘導されませんが,さらに次のS期で強く誘導されるということも明らかにしています。これらの結果から,DNMT1阻害で生じたヘミメチル化DNAが,次の細胞周期のS期で複製フォークと衝突を起こすことでDNA損傷が誘導されるというヘミメチル化起源モデルを私たちは提唱しています。ヘミメチル化DNAとNP95/UHRF1の複合体が複製フォークと衝突を起こすことでDNA損傷の原因になっている可能性も考えられ,本研究ではこれらのモデルを検証します。
主な論文
- Miura, H., Takahashi, S., Shibata, T., Nagao, K., Obuse, C., Okumura, K., Ogata, M., Hiratani, I., *Takebayashi, S. I. Mapping replication timing domains genome wide in single mammalian cells with single-cell DNA replication sequencing (scRepli-seq). Nat Protoc (in press).
- Takahashi, S., Miura, H., Shibata, T., Nagao, K., Okumura, K., Ogata, M., Obuse, C., *Takebayashi, S. I., and Hiratani, I. Genome-wide stability of the DNA replication program in single mammalian cells. Nat Genet 51:529-540 (2019)
- *Takebayashi, S., Ogata, M., and Okumura, K. Anatomy of Mammalian Replication Domains. Genes (Basel) 8: E110 (2017)
- Takebayashi, S., Tamura, T., Matsuoka, C., and Okano, M. Major and essential role for DNA methylation mark in mouse embryogenesis and stable association of Dnmt1 with newly replicated regions. Mol. Cell Biol 27:8243-8258 (2007)
- Sharif, J., Muto, M., Takebayashi, S., Suetake, I., Iwamatsu, A., Endo, T. A., Shinga, J., Mizutani-Koseki, Y., Toyoda, T., Okamura, K., Tajima, S., Mitsuya, K., Okano, M., and Koseki, H. The SRA protein Np95 mediates epigenetic inheritance by recruiting Dnmt1 to methylated DNA. Nature 450:908-912 (2007)
