研究課題名
非ゲノム情報による複製開始点制御の解明
研究内容
真核生物のモデル生物代表選手である出芽酵母においては、複製開始点がコンセンサス配列に規定されており、染色体上全ての複製開始点が同定されています。一方、ヒト細胞を含む哺乳動物の複製開始点には明確なコンセンサス配列が存在せず、非ゲノム情報により制御されていることが知られています(図A)。これは、初期発生等ダイナミックに染色体が変化する状況では、コンセンサス配列できっちり複製開始点を決めないことが有利だったことを示しているのかもしれません。この性質のため、ヒト細胞においてはいくつかの代表的な複製開始領域を除けば、全ゲノムにおける詳細な複製開始点の位置や各々の活性は理解されておらず、従って非ゲノム情報との詳細な関連も明らかになっていません。
DNA複製においてはリーディング鎖が連続して合成されるのに対し、ラギング鎖として非連続的に合成される岡崎フラグメントは、後にDNAリガーゼによって連結されます。そこで、DNAリガーゼを阻害し、岡崎フラグメントを集めて方向性を特定したままシーケンスすれば、染色体上の複製開始点の位置と活性を網羅的に同定できます(図B)。このOkazaki sequence(OK-seq)法は、出芽酵母のDNAリガーゼ温度感受性変異株を用いておこなわれましたが、ヒト細胞においてはDNAリガーゼを効率よく不活化する方法が無かったため、おこなわれたことがありません。そこで、私たちの研究室で開発したオーキシンデグロン法をヒト細胞のDNAリガーゼLIG1とLIG3に応用し、リガンド添加によりLIG1とLIG3を数時間以内に分解除去することが可能なヒト細胞を作出しました(図C)。本研究では、ヒト細胞を材料としたOK-seqにより、染色体上の複製開始点の位置と活性を調べるとともに、ChIP-seq等で調べられた非ゲノム情報との関連性を明らかにします。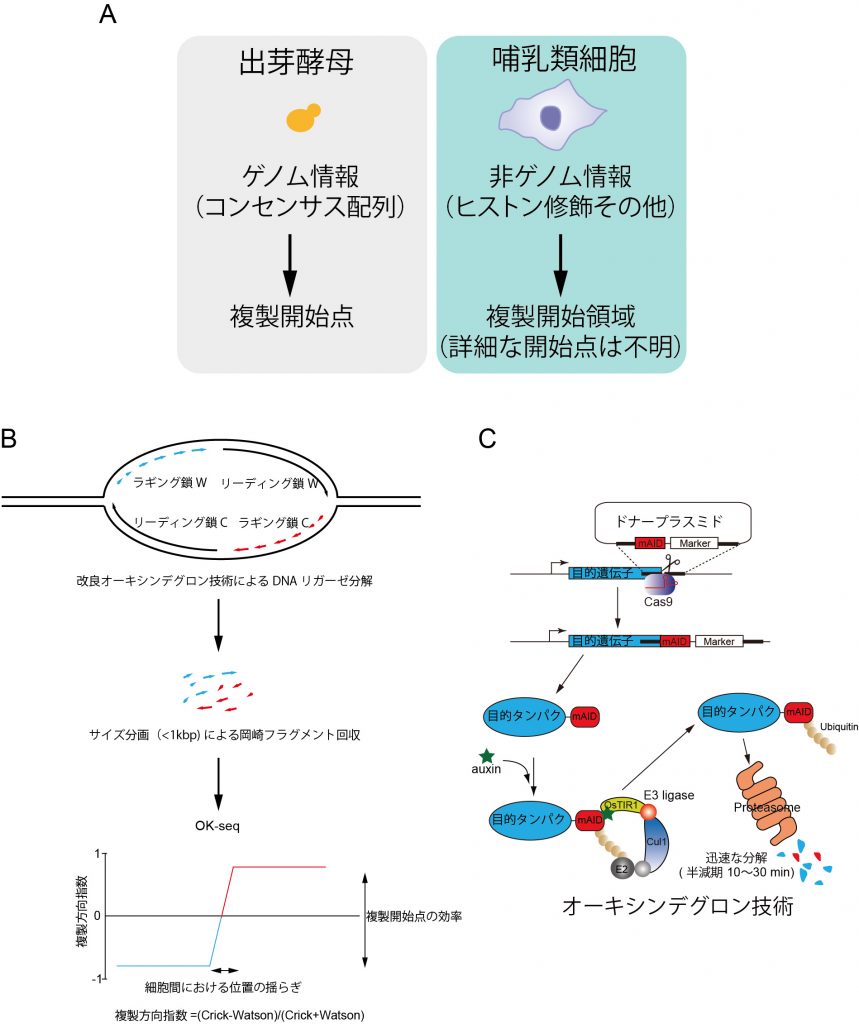
主な論文
- Natsume, T., Nishimura, K., Minocherhomji, S., Bhowmick, R., Hickson, ID., *Kanemaki, MT. Acute inactivation of the replicative helicase in human cells triggers MCM8-9-dependent DNA synthesis. Genes Dev 31, 816-829 (2017)
- Natsume, T., Kiyomitsu, T., Saga, Y., *Kanemaki, MT. Rapid protein depletion in human cells by auxin-inducible degron tagging with short homology donors. Cell Rep 15, 210-218 (2016)
- Nishimura, K., Ishiai, M., Horikawa, K., Fukagawa, T., Takata, M., Takisawa, H., *Kanemaki, MT. Mcm8 and Mcm9 form a complex that functions in homologous recombination repair induced by DNA interstrand crosslinks. Mol Cell 47, 511-522 (2012)
- Watase, G., Takisawa, H., *Kanemaki, MT. Mcm10 plays a role in functioning of the eukaryotic replicative DNA helicase, Cdc45-Mcm-GINS. Curr Biol 22, 343-349 (2012)
- Nishimura, K., Fukagawa, T., Takisawa, H., Kakimoto, T., *Kanemaki, M. An auxin-based degron system for the rapid depletion of proteins in nonplant cells. Nat Meth 6, 917-922 (2009)
