DNAメチル化によるゲノム情報安定化機構の解明
研究代表者
研究分担者
 鵜木 元香
鵜木 元香
Motoko Unoki
東京大学 人類遺伝学分野・准教授
 藤 泰子
藤 泰子Taiko To
東京工業大学 生命理工学院・准教授
研究内容
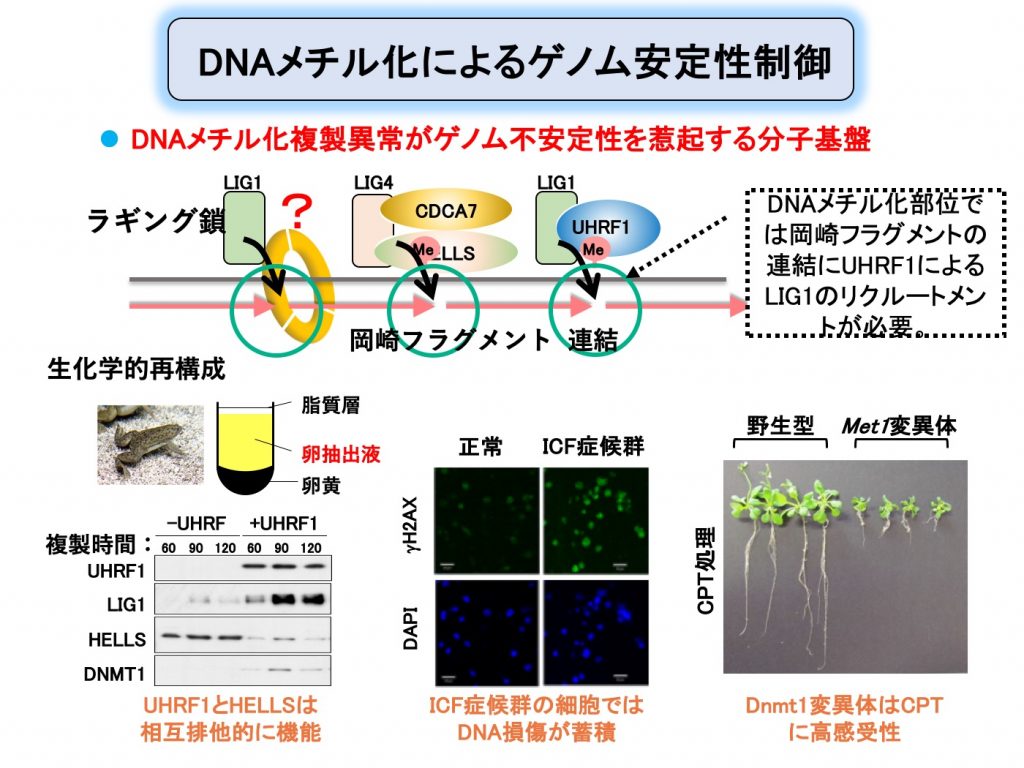
近年のがんなどのゲノム不安定疾患や、遺伝子改変動物の解析から、DNAメチル化複製機構が上位でゲノム複製や安定性維持を制御することが明らかとなっているが、その分子基盤は全くわかっていない。我々はこれまで、DNAメチル化とDNA鎖の共役した複製機構について、その全貌を分子構造学的に明らかにしてきた。ごく最近、DNA複製後に生じたヘミメチル化DNAにより集積したUHRF1が、DNMT1のみならず岡崎フラグメント連結に関わるLIG1の集積も制御していることが示された。さらに、シロイヌナズナを用いた遺伝学的解析からも、UHRF1変異体がDNA複製ストレスに対して選択的に感受性を示すことが分かった。これらの結果はDNAメチル化複製が岡崎フラグメントの連結制御を介してゲノム安定性に重要な役割を果たすことを強く示唆している。一方、哺乳類において受精直後の初期発生段階でゲノムワイドな受動的DNA脱メチル化が起こるが、その分子機構やゲノム情報維持における役割はほとんど解明されていない。我々はツメガエル卵抽出液を用いた無細胞生化学的手法を用いて、受動的DNA脱メチル化の再構成実験系を樹立した。以上の知見や解析技術を基に、本研究ではDNAメチル化複製機構によるゲノム安定性維持機構の本体を理解することを目的とする。具体的には、UHRF1依存的なLIG1リクルートの分子機構、UHRF1非存在下において生じる岡崎フラグメントの連結異常について解析を行い、DNAメチル化不全がゲノム不安定性をもたらす仕組みを明らかにする。また、遺伝性ゲノム不安定疾患であるICF細胞を用いて、DNAメチル化複製制御によるゲノム安定化機構を解析する。研究期間の後半においてはゲノムワイドな受動的DNA脱メチル化に焦点をあて、その分子機構を明らかにするとともに、受精卵の初期発生過程におけるゲノム安定性維持に果たす役割を明らかにする。
主な論文
中西真
- Nishiyama A., *Nakanishi M. Navigating the DNA methylation landscape of cancer. Trends Genet. 37, 1012-1027. (2021)
- Kumamoto S., Nishiyam, A., Chiba Y., Miyashita R., Konishi C., Azuma Y., *Nakanishi M. HPF1-dependent PARP activation promotes LIG3-XRCC1-mediated backup pathway of Okazaki fragment ligation. Nucleic Acids Res. 49, 5003-5016 (2021)
- Suzuki N., Johmura Y., Wang TW., Migita T., Wu W., Noguchi R., Yamaguchi K., Furukawa Y., Nakamura S., Miyoshi I., Yoshimori T., Ohta T., *Nakanishi M. TP53/p53-FBXO22-TFEB controls basal autophagy to govern hormesis. Autophagy. 17, 3776-3793 (2021)
- Johmura Y., et al., *Nakanishi M. (23人中23番目) Senolysis by glutaminolysis inhibition ameliorates various age-associated disorders. Science.371, 265-270 (2021)
- Omor, S., et al.,*Nakanishi M. (33人中33番目) Generation of a p16 Reporter Mouse and Its Use to Characterize and Target p16high Cells In Vivo. Cell Metab. 32, 814-828 (2020)
- Johmura Y., Harris AS., Ohta T., *Nakanishi M. FBXO22, an epigenetic multiplayer coordinating senescence, hormone signaling, and metastasis. Cancer Sci. 111, 2718-2725 (2020)
- Nishiyama, A., et al., *Nakanishi, M. (18人中18番目) Two distinct modes of DNMT1 recruitment ensure stable maintenance DNA methylation. Nature Commun 11, 1222 (2020)
- Nishimura, K., et al., *Nakanishi, M. (12人中12番目) Cdk1-mediated DIAPH1 phosphorylation maintains metaphase cortical tension and inactivates the spindle assembly checkpoint at anaphase. Nature Commun 10, 981 (2019)
- Johmura, Y., et al., *Nakanishi, M. (20人中20番目) Fbxo22-mediated KDM4B degradation determines selective estrogen receptor modulator activity in breast cancer. J Clin Invest 128, 5603-5619 (2018)
- Ishiyama, S., et al., *Nakanishi, M. (22人中22番目) Structure of the DNMT1 reader module complexed with a unique two-mono-ubiquitin mark on histone H3 reveals the basis for DNA methylation maintenance. Mol Cell 68, 350-360 (2017)
- Shimada, M., et al., *Nakanishi, M. (10人中10番目) Essential role of autoactivation circuitry on Aurora B-mediated H2AX-pS121 in mitosis. Nature Commun 7, 12059 (2016)
- Johmura, Y., et al., *Nakanishi, M. (15人中15番目) SCFFbxo22-KDM4A targets methylated p53 for degradation and regulates senescence. Nature Commun 7, 10574 (2016)
- Johmura, Y., et al., *Nakanishi, M. (12人中12番目) Necessary and sufficient role for a mitosis skip in senescence induction. Mol Cell 55, 73-84 (2014)
- Hirokawa, T., et al., *Nakanishi, M. (9人中9番目) CBP-93872 inhibits NBS1-mediated ATR activation, abrogating maintenance of the DNA double-strand break-specific G2 checkpoint. Cancer Res 74, 3880-3889 (2014)
- Nishiyama, A., et al., *Nakanishi, M. (12人中12番目) Uhrf1-dependent H3K23 ubiquitylation couples maintenance DNA methylation and replication. Nature 502, 249-253 (2013)
- Delhase, M., et al., Nakanishi, M. (13人中13番目) TANK-binding kinase 1 (TBK1) controls cell survival through PAI-2/serpinB2 and glutaminase 2. Proc Natl Acad Sci 109, E177-186 (2012)
- Niida, H., et al., *Nakanishi, M. (17人中17番目) Cooperative functions of Chk1 and Chk2 reduce tumor susceptibility in vivo. EMBO J 29, 3558-3570 (2010)
- Niida, H., et al., *Nakanishi, M. (12人中12番目) Essential role of Tip60-dependent recruitment of ribonucleotide reductase at DNA damage sites in DNA repair during G1 phase. Genes Dev 24, 333-338 (2010)
- Shimada, M., et al., *Nakanishi, M. (5人中5番目) PP1g is a phosphatase responsible for dephosphorylation of histone H3 at threonine 11 after DNA damage. EMBO Rep 11, 663-889 (2010)
- Katsuno, Y., et al., *Nakanishi, M. (10人中10番目) Cyclin A-Cdk1 regulates the origin firing program in mammalian cells. Proc Natl Acad Sci 106, 3184-3189 (2009)
- Shimada, M., et al., *Nakanishi, M. (7人中7番目) Chk1 is a histone H3 threonine 11 kinase that regulates DNA damage-induced transcriptional repression. Cell 132, 221-232 (2008)
- Tojima, Y., et al., *Nakanishi, M. (12人中12番目) NAK is an IkB kinase-activating kinase. Nature 404, 778-782 (2000)
鵜木元香
- Unoki, M., et al., (7人中1番目)CDCA7 and HELLS suppress DNA:RNA hybrid-associated DNA damage at pericentromeric repeats. Sci Rep 10, 17865 (2020)
- *Unoki, M., et al., (5人中1番目) CDCA7 and HELLS mutations undermine non-homologous end joining in centromeric instability syndrome, J Clin Invest 129, 78-92 (2019)
- Maenohara, S., *Unoki, M., et al., (5人中2番目) Role of UHRF1 in de novo DNA methylation in oocytes and maintenance methylation in preimplantation embryos. PLoS Genet 13, e1007042 (2017)
- Thijssen, P.E., Unoki, M., et al., (29人中7番目) Mutations in CDCA7 and HELLS cause immunodeficiency-centromeric instability-facial anomalies syndrome. Nature Commun 6, 7870 (2015)
- *Unoki, M., et al., (12人中1番目) Lysyl 5-hydroxylation, a novel histone modification, by Jumonji domain containing 6 (JMJD6). J Biol Chem 288, 6053-6062 (2013)
藤泰子
- To, T.K., et al., (9人中1番目) RNA interference-independent reprogramming of DNA methylation in Arabidopsis. Nature Plants 6, 1455–1467 (2020)
- *Kim, J.M., To, T.K., et al., * Seki, M. (30人中22番目) Acetate-mediated novel survival strategy against drought in plants. Nature Plants 3, 17097 (2017)
- Ito, T., Tarutani, Y., To, T.K. , et al., *Kakutani, T. (12人中3番目) Genome-wide negative feedback drives transgenerational DNA methylation dynamics in Arabidopsis. PLoS Genet 11, e1005154 (2015)
- To, T.K., et al., *Kakutani, T. (3人中1番目) DNA methylation within transcribed regions. Plant Physiol 168, 1219-1225 (2015)
- *To, T.K., Kim, J.M. (2人中1番目) Epigenetic regulation of gene responsiveness in Arabidopsis. Front Plant Sci 4, 548 (2014)