DNAメチル化とH3K9me3の確立と維持の構造基盤
研究内容
DNA メチル化やヒストン H3K9 のメチル化(H3K9me3)は、クロマチンの構造変換を制御する遺伝子発現抑制的な非ゲノム情報である。これらの非ゲノム情報が発生・分化過程でどのように確立し、そして多細胞形質体を維持するために体細胞分裂後にどのように複製されていくのかは不明な点が多い。本研究では、非ゲノム情報による遺伝子発現抑制の確立と複製に関わる構造基盤として、ユビキチン化された複製因子 PAF15によるDNA維持メチル化、UHRF1によるユビキチン修飾の確立、 SETDB1によるH3K9me3の確立に注目し、それぞれの分子機構を構造生物学的な観点から明らかにする。
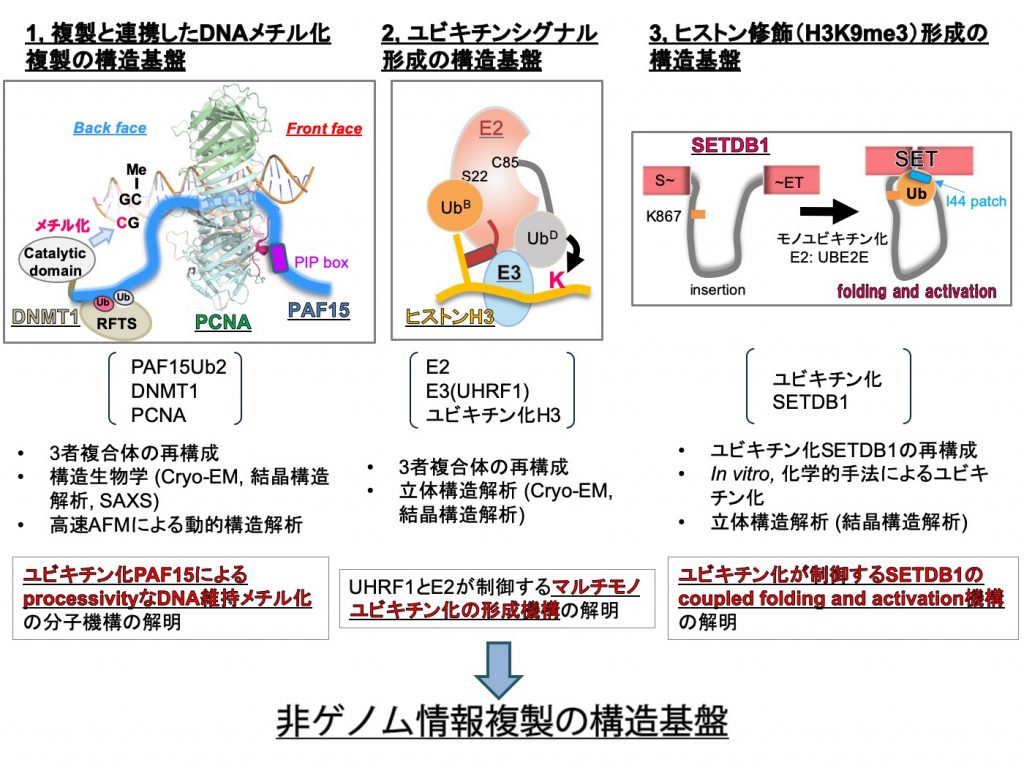
【研究項目1】 ユビキチン化 PAF15 による processivity な DNA 維持メチル化機構の解明
DNA クランプである PCNA と相互作用する複製因子 PAF15は、DNA 複製と連携して DNA維持メチル化を制御する新たな因子である。K15とK24がユビキチン化されたPAF15(PAF15Ub2)は、DNA 維持メチル化酵素 DNMT1 を複製サイトに呼び込む。PCNA:PAF15Ub2:DNMT1 複合体構造をクライオ電子顕微鏡による単粒子解析で決定し、複製と連携したDNA 維持メチル化の新たな分子機構の解明を目指す。
【研究項目2】 UHRF1によるマルチモノユビキチン修飾の形成機構の解明
DNA維持メチル化ではDNMT1はユビキチン化H3やPAF15によってDNAメチル化サイトに呼び込まれる。しかし、UHRF1が触媒するヒストンH3やPAF15のマルチモノユビキチン修飾の形成機構は不明である。本項目では、UHRF1:E2酵素:基質複合体をX線結晶構造解析やクライオ電子顕微鏡で解明し、UHRF1が触媒するマルチモノユビキチン修飾の形成機構の解明を目指す。
【研究項目3】ユビキチン化 SETDB1 によるヒストン H3K9me3 の確立・複製機構の解明:
H3K9me3 の確立・複製を行う SETDB1 は、SET ドメイン内の insertion loop のモノユビキチン化が、SET ドメインの構造形成(folding)と活性に重要である。ユビキチン化 SETDB1 の立体構造を X線結晶構造解析で決定し、ユビキチン化による SETDB1 の Coupled Folding and Activation 機構を解明すし、ユビキチン化と連携したヒストン H3K9me3 の確立・複製機構を解明する。
主な論文
- Kori S., et al., Arita K. (13人中13番目) Structure-based screening combined with computational and biochemical analyses identified the inhibitor targeting the binding of DNA Ligase 1 to UHRF1. Bioorganic & Medicinal Chemistry. 52, 116500-116500 (2021)
- Funyu T., Kanemaru Y., Onoda H., Arita K. Preparation of the ubiquitination-triggered active form of SETDB1 in E. coli for biochemical and structural analyses. The Journal of Biochemistry. 170, 655-662 (2021)
- Ohno A., et al., (14人中5番目) Crystal structure of inhibitor-bound human MSPL that can activate high pathogenic avian influenza. Life Sci Alliance. 4, e202000849 (2021)
- Furukawa A., Walinda E., Arita K., Sugase K. Structural dynamics of double-stranded DNA with epigenome modification. Nucleic Acids Research. 49, 1152–1162 (2021)
- Kori S., et al., Arita K. (9人中9番目) Serine 298 phosphorylation in linker 2 of UHRF1 regulates ligand-binding property of its tandem Tudor domain. Mol. Biol. 432, 4061-4075 (2020)
- Nishiyama A., et al., Arita K. (18人中16番目) Two distinct modes of DNMT1 recruitment ensure stable maintenance DNA methylation. Nature Commun 11, 1222 (2020)
- Jimenji T., et al., Arita K. (4人中4番目) Structure of PCNA in complex with DNMT1 PIP box reveals the basis for the molecular mechanism of the interaction. BBRC 516, 578-583 (2019)
- Kori S., et al., Arita K. (14人中14番目) Structure of the UHRF1 Tandem Tudor Domain bound to a methylated non-histone protein, LIG1, reveals rules for binding and regulation. Structure 27, 485-496 (2019)
- Ishiyama S., et al., Arita K. (22人中21番) Structure of the Dnmt1 reader module complexed with a unique two-mono-ubiquitin mark on histone H3 reveals the basis for DNA methylation maintenance. Mol Cell 68, 350-360 (2017)
- Ferry L., et al., Arita K. (21人中19番目) Methylation of DNA Ligase 1 by G9a/GLP recruits UHRF1 to replicating DNA and regulates DNA methylation. Mol Cell 67, 550-565 (2017)
- Morimoto D., et al., Arita K. (17人中10番目) The unexpected role of polyubiquitin chains in the formation of fibrillar aggregates. Nat Commun. 6, (2015)
- Berkyurek AC., et al., Arita K. (7人中3番目) The DNA Methyltransferase Dnmt1 Directly Interacts with the SET and RING Finger Associated (SRA) Domain of the Multifunctional Protein Uhrf1 to Facilitate Accession of the Catalytic Center to Hemi-methylated DNA. Biol. Chem 289, 379-386 (2014)
- Unoki M., et al., Arita K. (12人中4番目) Lysyl 5-Hydroxylation, a Novel Histone Modification, by Jumonji Domain Containing 6 (JMJD6) J. Chem. 288, 6053-6062 (2013)
- Otani J., et al., Arita K. (9番中2番目) Structural basis of the versatile DNA recognition ability of the methyl CpG binding domain of methyl-CpG binding domain protein 4. Biol. Chem. 288, 6351-6362 (2013)
- Nishiyama A., et al., Arita K. (12人中8番目) Uhrf1-dependent ubiquitylation of histone H3 at lysine 23 couples maintenance DNA methylation and DNA replication. Nature 502, 249-253 (2013)
- Arita K., et al., Shirakawa M. (12人中1番目) Recognition of modification status on a histone H3 tail by linked histone reader modules of the epigenetic regulator UHRF1. 109, 12950-12955 (2012)
- Otani J., et al., Arita K. (6人中3番目) Structural basis for recognition of H3K4 methylation status by the DNMT3A ADD domain. EMBO Rep. 11, 1235-1241 (2009)
- Arita K., et al., (5人中1番目) Recognition of hemi-methylated DNA by the SRA protein UHRF1 by a base-flipping mechanism. Nature 455, 818-821 (2008)
- Arita K. et al., (6人中1番目) Structural basis for histone N-terminal recognition by human peptidylarginine deiminase 4. PNAS. 103, 5291-5296 (2006)
- Arita K., et al., (6人中1人目) Structure basis for Ca2+-induced activation of human PAD4. 11, 777-783 (2004)
 有田 恭平
有田 恭平