[研究成果] 鵜木(中西班)と有田班の共同研究がHum Mol Genet誌に掲載されました!
2022.12.03
Unoki M, Velasco G, Kori S, Arita K, Daigaku Y, Fujimoto A, Ohashi H, Kubota T, Miyake K, Sasaki H. Novel compound heterozygous mutations in UHRF1 are associated with atypical immunodeficiency, centromeric instability, and facial anomalies syndrome with distinctive genome-wide DNA hypomethylation. Hum. Mol. Genet. 2023 Apr 20;32(9):1439-1456. doi: 10.1093/hmg/ddac291.
DNAメチル化異常を伴う先天性疾患ICF症候群の新たな原因遺伝子としてUHRF1を同定
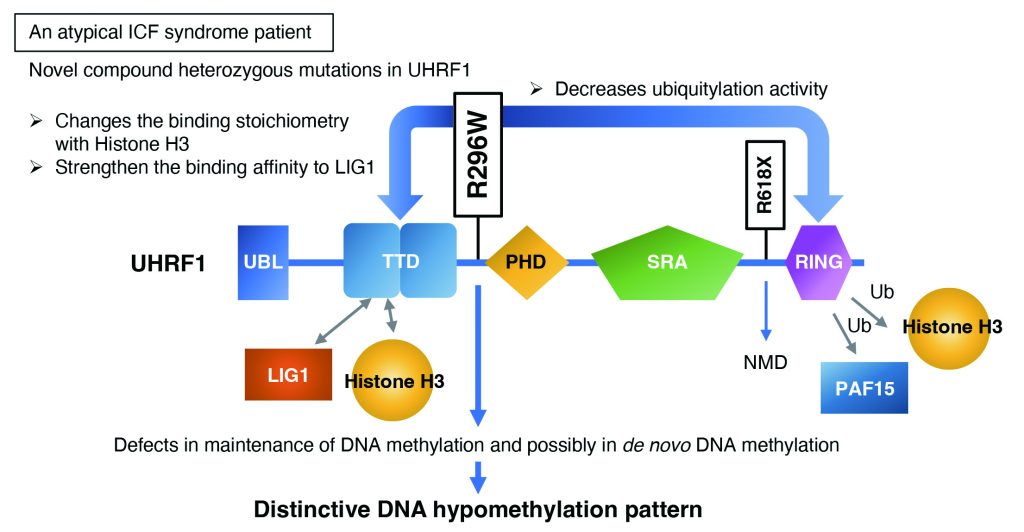
Immunodeficiency centromeric instability facial anomalies(ICF)症候群患者は、ほとんどの症例で、DNMT3B、ZBTB24、CDCA7、HELLSのいずれかの遺伝子に変異を有しているが、少数のICF患者の原因遺伝子は依然不明であった(CDCA7とHELLSは、私たちが2015年に変異を報告した遺伝子である:Thijssen et al., Nat Commun, 2015)。今回私たちは、このようなICF患者のうち1名(患者ID:P4)が、UHRF1遺伝子に、これまで報告されていない複合ヘテロ接合体性変異(R296WとR618X)を有していることを突き止めた。UHRF1(別名ICBP90)は2004年に私がDNAメチル化を認識する新規蛋白質として同定して以来(Unoki et al., Oncogene, 2004)、本新学術グループのメンバーである古関先生、有田先生、中西班の西山先生らによって詳細な機能が解明されてきた維持DNAメチル化に必須の蛋白質である。これまでUHRF1の変異は、マルチローカスインプリンティング異常症の1症例でのみ報告があり(変異と疾患の因果関係は不明)、本報告が、変異がUHRF1の機能に及ぼす影響まで詳細に調べた世界初の報告となる。今回私たちが見つけたR618X変異は、UHRF1のSRAとRINGドメイン間のリンカー領域にあり、患者細胞ではこの変異を有するUHRF1蛋白質の発現が認められなかったことから、当該変異はナンセンス変異依存mRNA分解を引き起こすと考えられた。R296W変異はタンデムTudorドメイン(TTD)とプラントホメオドメイン(PHD)間のリンカー領域に位置しており、生化学的解析の結果、当該変異は、UHRF1とヒストンH3との結合比を1:1から1:2へと変化させ、またLIG1との結合親和性を強めることがわかった。一方、R296W変異はUHRF1のユビキチン化基質であるヒストンH3およびPAF15に対するユビキチン化活性を低下させることがわかった。ゲノムワイドなDNAメチル化解析の結果、当該患者はICF症候群の主徴であるセントロメア・ペリセントロメア反復配列の低メチル化を有することがわかった。しかしながら、この患者は、他のICF症候群患者(1型、2型、3型、4型)と比較して、特徴的な低メチル化パターンを有することもわかった。私たちは、この患者のモデル細胞株を樹立し、R296W変異がペリセントロメア反復配列の低メチル化を引き起こすことを確認し、この変異がhypomorphicな変異であることを証明した。UHRF1とLIG1、PAF15、ヒストンH3との適切な相互作用は維持DNAメチル化のプロセスに必須であるため、R296W変異は当該プロセスの効率を低下させる可能性がある。さらにR296W変異はUHRF1とde novoDNAメチル化酵素であるDNMT3Bとの結合親和性を弱めたことから、発生過程におけるde novo DNAメチル化にも影響する可能性がある。今回の報告は、DNAメチル化の制御におけるUHRF1の役割についての理解をより深めるものである。