[研究成果] 京都大学と横浜市大(有田班他)の共同研究成果がNucleic Acids Res誌に掲載されました!
2020.12.23
Furukawa A, Walinda E, Arita K, Sugase K. Structural dynamics of double-stranded DNA with epigenome modification. Nucleic Acids Res 2020 Dec 18. in press.
プレスリリースはこちら。
メチル基1つでDNAの運動性が変わることを解明
―運動性というDNA上の目印―
概要
横浜市立大学大学院生命医科学研究科 古川亜矢子 特任助教(研究開始時:サントリー生命科学財団 研究員)、京都大学大学院医学研究科 Erik Walinda 助教、横浜市立大学大学院生命医科学研究科 有田恭平 准教授、京都大学大学院工学研究科 菅瀬謙治 准教授(研究開始時:サントリー生命科学財団 グループ長)の研究グループは、ゲノムの安定性や遺伝子発現の制御などに関わるDNAのエピゲノム修飾が二本鎖DNAの局所的な運動性を大きく変化させることを明らかにしました。
DNAのシトシンはエピゲノム修飾の一つとしてメチル化されますが、この修飾によりDNAの二重らせん構造はほとんど変化しません。しかし、ある種のタンパク質はメチル化されたシトシンを特異的に認識します。このことは大きな謎でした。本研究では、たった一つのメチル基が二本鎖DNAに導入されるだけでその運動性が局所的に大きくなることをNMR(核磁気共鳴)と分子動力学計算によって明らかにしました。このことは運動性の違いがエピゲノム修飾の目印となっていることを示唆します。本研究の成果は、将来的に「エピゲノム修飾はDNAを化学的に修飾するだけでなく運動性も修飾する性質がある」という概念の刷新に繋がることが期待されます。
本成果は、2020年12月19日に国際学術誌「Nucleic Acid Research」にオンライン掲載されました。
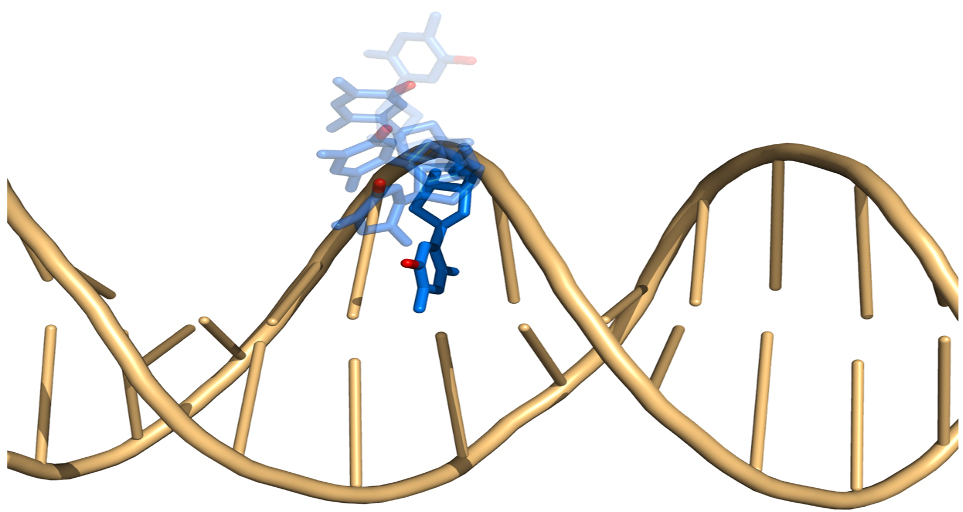
図:二本鎖DNAにたった一つメチル基が入ると局所的な運動性が大きくなることを明らかにしました。メチル化されたシトシンを青色の原子構造(メチル基は赤色)で他は金色のリボンモデルで表示。
1.背景
エピゲノム注1修飾の一つであるシトシンの修飾は、ゲノムの安定性や遺伝子発現の制御などにおいて重要な役割を果たします。この制御の破綻は細胞のガン化にも繋がります。二本鎖DNA中のCG配列(相補鎖はGC配列;Cはシトシン、Gはグアニン)のシトシンがDNAメチル化酵素によってメチル化されると、シトシンは5-メチルシトシンになります(図1)。さらに、5-メチルシトシンはTET酵素によって5-ヒドロキシメチルシトシン、5-ホルミルシトシン、5-カルボキシシトシンへと変化します。
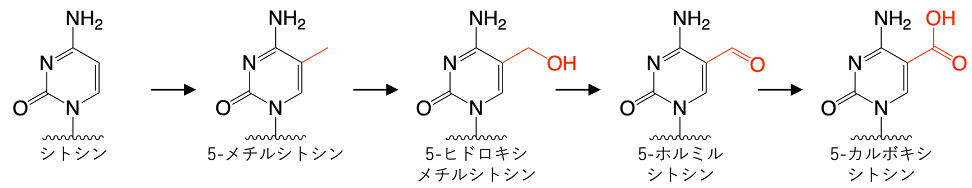
図1.シトシンのエピゲノム修飾
これらの修飾シトシンは様々なタンパク質によって特異的に認識されます。その中の1つであるユビキチンリガーゼUHRF1は、CG配列とGC配列の片側だけがメチル化(ヘミメチル化)された二本鎖DNAを認識しますが、CG配列とGC配列の両者がメチル化された二本鎖DNAや未修飾の二本鎖DNAは認識しません。過去に、共同研究者の有田准教授らによってヘミメチル化二本鎖DNAとUHRF1の複合体の結晶構造が決定され、5-メチルシトシンは二重らせん構造の外に露出した状態でUHRF1に認識されることが明らかにされました(図2)。しかし、この結晶構造からはUHRF1がどのようにしてヘミメチル化二本鎖DNA中の5-メチルシトシンだけを外に露出させ認識するのかは分かりませんでした。我々はUHRF1がヘミメチル化二本鎖DNAを特異的に認識する理由として、シトシンのメチル化によって二本鎖DNAの運動性が変化し、それがUHRF1に対する目印になっているのではないかと考えました。
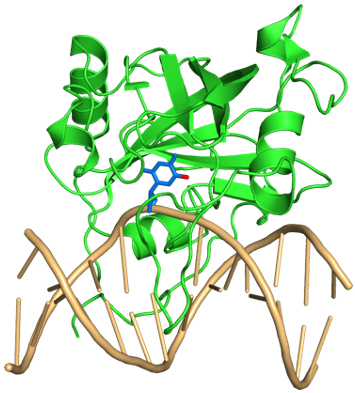
図2.ヘミメチル化二本鎖DNAとUHRF1の複合体構造
2.研究手法・成果
二本鎖DNAは、アデニン-チミンとグアニン-シトシンのワトソン-クリッ ク型塩基対を形成しますが(図3)、塩基対は常に安定ではなく過渡的に開いた状態にもなります。塩基対が開いて元に戻る(閉じる)過程はマイクロ秒オーダーの極めて速いタイムスケールで起こるため、その定量的な解析は困難でした。本研究では二本鎖DNA中のイミノプロトン(図3)と水との交換速度を決定するCLEANEX-PM法と緩和分散法注2と呼ばれる2つのNMR法注3を駆使して、塩基対が開く速度(kopen)と閉じる速度(kclose)を半定量的に明らかにしました。異なるシトシン修飾を含む6種類の二本鎖DNAのkopenとkcloseを計測した結果、ヘミメチル化二本鎖DNAの場合にだけkopenとkcloseの増加が見られ(図4)、他のシトシン修飾ではkcloseのみが増加しました。kopenの増加はワトソン-クリック型塩基対が不安定化したことを表し、kcloseの増加は塩基対が開 いた状態の寿命が短くなったことを表します。次に、NMRで計測できるタイムスケールよりも速いナノ秒オーダーの運動性を調べるために分子動力学計算注4を実施しました。その結果、二本鎖DNAの局所的な曲率や柔軟性がシトシン修飾によって変化することが分かりました。以上のことから、シトシン修飾によって二本鎖DNAの局所的な運動性がナノ秒からマイクロ秒のタイムスケールで変化し、とくに片方のDNA鎖だけがメチル化されることが最も顕著な変化を引き起こすことが分かりました。すなわち、たった一つのメチル基が二本鎖DNAに入ることによって、その運動性が大きく変わるということです。
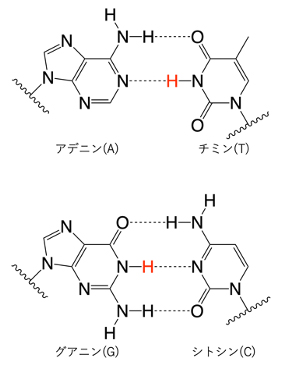
図3.DNAの塩基対
イミノプロトン(赤)

図4.ヘミメチル化によって変化する二本鎖DNAの運動性
CLEANEX-PMによりイミノプロトンと水の交換速度k1Hが求まり、緩和分散法によりkex(=kopen+kclose)が求まる。本実験の条件では、kopen∝k1Hとkclose≈kexが成り立つ。
3.波及効果、今後の予定
タンパク質がエピゲノム修飾されたDNAを認識する際に、タンパク質側が柔軟に構造変化することは比較的よく知られていることですが、本研究によりDNA側も実は柔軟な構造をしており、それがエピゲノム修飾の種類や位置に依存して局所的に変化することが解明されました。一般にDNAのエピゲノム修飾はメチル化やヒドロキシメチル化といった化学的な修飾と考えられていますが、本研究成果が契機となり、将来的に「エピゲノム修飾にはDNAの運動性を修飾する役割もある」という新しい概念が確立されることが期待されます。この概念を突き詰めると、DNAを認識するタンパク質は二本鎖DNAのわずかな化学構造の違いだけでなく、その運動性の違いも認識しているという仮説の立脚に繋がります。今後はこの仮説を立証するために、UHRF1などエピゲノム修飾を認識するタンパク質の存在下で、本研究と同様な二本鎖DNAの運動性の解析を実施する予定です。
4.研究プロジェクトについて
本研究は、菅瀬と古川がサントリー生命科学財団に所属時に始まり、それぞれが現職に就いてからは京都大学と横浜市立大学との共同研究として実施しました。サントリー生命科学財団、科学研究費補助金、国立研究開発法人日本医療研究開発機構(AMED)創薬等ライフサイエンス研究支援基盤事業 創薬等先端技術支援基盤プラットフォーム(BINDS)の支援を受けました。
<用語解説>
注1 DNAの塩基配列は変えることなくDNAやヒストンの化学修飾などによって遺伝子のはたらきを制御する仕組み。
注2 マイクロ秒からミリ秒オーダーの構造変化を定量的に解析するNMR法。
注3 磁場中に置かれた試料に含まれる水素原子などの核スピンの共鳴現象を観測することによって試料の構造や運動性などを原子レベルで解析するための装置。
注4 分子の動きをコンピュータで予測するシミュレーション法
<研究者のコメント>
結晶構造解析によって多くのDNAの立体構造が決定されていますが、それらは全て静止した構造です。教科書にもそのようなイメージで描かれるため、多くの人はDNAの構造は硬いものと見ています。しかし、本研究で明らかにしたようにDNAの構造は非常に柔軟であります。そのため本プレスリリースでは、DNAは柔軟な分子でかつその柔軟性が変わりうるというメッセージを一般の人にも届けたいと思います。
<論文タイトルと著者>
タイトル:Structural dynamics of double-stranded DNA with epigenome modification
(エピゲノム修飾を含む2本鎖DNAのダイナミクス構造)
著 者:Ayako Furukawa, Erik Walinda, Kyohei Arita, and Kenji Sugase
掲 載 誌: Nucleic Acid Research DOI:10.1093/nar/gkaa1210
<お問い合わせ先>
<研究に関するお問い合わせ>
菅瀬 謙治 (すがせけんじ)
京都大学大学院工学研究科分子工学専攻・准教授
TEL:090-5897-5580
FAX:075-383-2541
E-mail:sugase@moleng.kyoto-u.ac.jp
<報道に関するお問い合わせ>
京都大学総務部広報課 国際広報室
TEL:075-753-5729 FAX:075-753-2094
E-mail:comms@mail2.adm.kyoto-u.ac.jp
横浜市立大学 研究・産学連携推進課 研究企画担当
TEL:045-787-2527 FAX:045-787-2509
E-mail:kenkyupr@yokohama-cu.ac.jp