クロマチン構造が規定する分化時の段階的遺伝子発現制御機構の解明
研究内容
細胞分化に必要な選択的遺伝子発現は、クロマチンの凝集と弛緩に至る幅広いクロマチン構造制御が関与する。研究代表者はこれまで、計算機により新規マウスヒストンバリアント群を網羅的に同定し(Epigenetics & Chromatin, 2015)、以来、クロマチン組成が規定する遺伝子の選択的発現機構の解明に取り組んできた。特にクロマチン組成による遺伝子発現制御のメカニズムを解明するため、ウェットとデータ解析技術の開発を同時に進めている。高感度な少数細胞エピゲノム解析技術ChIL-seqを開発し、計測技術開発が進んだ一方、単一細胞レベルのエピゲノムデータを用いる細胞分化軌跡推定は、スタンダードな解析手法が確立されていない。
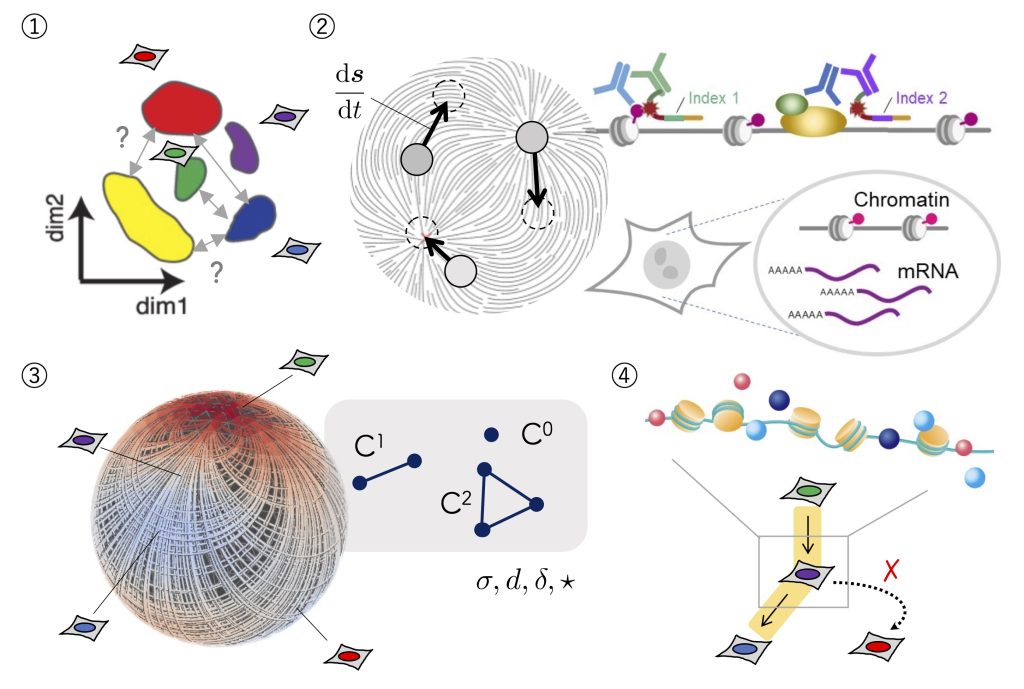
そこで本計画では、大規模オミクスデータによって明らかとなってきた細胞種や状態のクラスター同士を繋ぐ経路(図①;分化過程や状態遷移のギャップ)をクロマチン構造の視点から明らかにすることを目指す。まず、エピゲノムとトランスクリプトーム情報を単一細胞内で同時に取得するマルチオミクス計測法の確立を目指す(図②;状態変化を計測するマルチオミクス解析技術の確立)。次に、現在開発を続けている位相的データ解析手法を応用し、高次元な遺伝子発現空間のランドスケープを推定する(図③)。ランドスケープ上の分化経路を規定するクロマチン構成因子(図④)をマルチオミクスデータの解析から明らかにすることで、分化における段階的遺伝子発現制御機構の解明に迫る。
主な論文
1. Harada, A.†, Maehara, K†. et al.(17人中2番目), Histone H3. 3 sub-variant H3mm7 is required for normal skeletal muscle regeneration. Nature communications 9, 1–13 (2018).
2. Harada, A.†, Maehara, K† et al.(10人中2番目), A chromatin integration labelling method enables epigenomic profiling with lower input. Nature cell biology 21, 287–296 (2019).
3. Handa, T.†, Harada, A.†, Maehara, K.† et al.(9人中3番目), Chromatin integration labeling for mapping DNA-binding proteins and modifications with low input. Nature Protocols 15, 3334–3360 (2020).
4. Maehara, K. et al.(12人中1番目), Modeling population size independent tissue epigenomes by ChIL-seq with single thin sections. Molecular systems biology 17, e10323 (2021).
5. Fujii, T.†, Maehara, K.†*, Fujita, M. & Ohkawa*, Y.(4人中2番目), Discriminative feature of cells characterizes cell populations of interest by a small subset of genes. PLoS computational biology 17, e1009579 (2021).
(†: 共同筆頭著者, *: 共同責任著者)
